Moday, March 9th
|
10:00-10:45 Simon Cauchemez - Infectious diseases: Characterizing transmission from incomplete
epidemiological data
Abstract: In this presentation, I will give an overview of the challenges
we are confronted with when we try to estimate the transmission
properties of infectious diseases from epidemiological data, in a
context where data are often sparse, incomplete and biased. The talk
will be illustrated with a number of examples for different pathogens
(Ebola, chikungunya, MERS-CoV, influenza...).
|
11:00-11:45 Elisabeta Vergu - Analyse du réseau dynamique des mouvements de bovins en France et évaluation des risques associés de propagation d'agents pathogènes
Abstract: L'étude de la propagation d'agents pathogènes dans une métapopulation doit comporter l'investigation de la structure sous-jacente représentée par le réseau de contacts entre les différentes entités. Ici, nous nous intéressons au réseau orienté, pondéré et dynamique constitué par les mouvements commerciaux d'animaux entre troupeaux bovins en France. Les propriétés de ce réseau ont été explorées pour différentes granularités spatiales et fenêtres temporelles. Une catégorisation des troupeaux basée sur des considérations économiques a permis d'évaluer le risque lié à la propagation d'agents pathogènes et les conséquences en termes épidémiologiques et économiques, par des expériences de percolation et de calcul du "ratio d'accessibilité du réseau" prenant en compte les propriétés dynamiques de ce dernier.
|
11:45-12:30 Arnaud Nucit - Estimation statistique des paramètres d'intérêt d'un modèle épidémique
Abstract: Nous considérons deux types de modèles épidémiques paramétriques basés sur un taux de transmission constant ou variable et comparons différentes méthodes d'estimation. Nous utilisons des méthodes paramétriques et non-paramétriques pour aboutir finalement à l'estimation du taux de transmission de la maladie. Nous menons une étude par simulation pour comparer les qualités des différents estimateurs introduits et finissons par une application sur des données réelles : celles du virus informatique CodeRed et celles du VIH.
|
Lunch
|
14:30-15:15 Anthony Cousien - - Modélisation de la transmission du virus de l'hépatite dans une population d'usagers de drogues injectables
Abstract: L'hépatite C est une infection provoquée par le virus de l'hépatite C. Dans environ 75% des cas, l'infection devient chronique et est responsable d'une fibrose du foie pouvant conduire à terme à des complications dont la mortalité est élevée, comme la cirrhose ou le carcinome hépatocellulaire. En France, les nouvelles infections se déclarent majoritairement chez les usagers de drogues injectables (UDI), malgré les mesures de réduction des risques mises en place et la disponibilité de traitements de plus en plus efficaces.
L'objectif de ce travail est d'évaluer l'impact sur la transmission du virus et sur la mortalité associée à l'hépatite C de stratégies de réduction des risques et d'améliorations dans la cascade de soins dans une population d'UDI en France. Des projections pour l'incidence, la prévalence et le nombre de complications hépatiques ont été réalisées grâce à un modèle stochastique individu-centré prenant en compte le réseau social au moyen d'un graphe aléatoire. Les données nécessaires à l'implémentation du modèle étant peu nombreuses, les paramètres manquants ont été estimées à partir des données disponibles par calcul bayésien approché. Finalement, l'estimation de l'impact de l'incertitude sur les valeurs de certains paramètres par le calcul des indices de Sobol, couramment utilisés dans un cadre déterministe, pose problème dans le cadre d'un modèle stochastique.
|
15:30-16:15 Cyrielle Dumont - Planification adaptative pour des modèles non linéaires à effets mixtes : application en pharmacocinétique pédiatrique
Abstract: Les modèles non linéaires à effets mixtes (MNLEM) sont utilisés en pharmacocinétique de population pour l'analyse des concentrations plasmatiques de l'ensemble des patients lors du développement de médicament. Des approches basées sur la matrice d'information de Fisher (MF) peuvent être utilisées pour optimiser le protocole de ces études. Ces approches reposent sur de l'information a priori, qui peut être difficile à appréhender et constitue donc un facteur limitant. En effet, les données pharmacocinétiques issues de populations particulières (enfants, maladies rares,...) ne sont souvent pas disponibles. Pour pallier cette difficulté, une alternative est la planification adaptative, qui est de plus en plus développée pour les essais cliniques randomisés ou les études de recherche de dose, mais que très peu et récemment appliquée dans le cadre des MNLEM. Les objectifs de ce travail sont de développer l'optimisation du déterminant de MF pour des protocoles adaptatifs et d'évaluer, par une étude de simulation, l'impact des protocoles adaptatifs sur la précision d'estimation des paramètres quand les 'vrais' paramètres pharmacocinétiques sont différents des paramètres a priori. Les résultats mettent en évidence une moins bonne précision d'estimation des paramètres avec le protocole non adaptatif, obtenu à partir de paramètres a priori différents des 'vrais' paramètres. Le protocole adaptatif permet en partie de compenser ces imprécisions d'estimation.
|
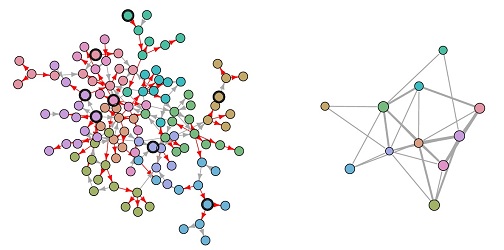
16:15-17:00 Fabrice Rossi -
Analyse exploratoire d'un graphe d'infection
Résumé : Nous étudions dans cette présentation un graphe d'infection par le VIH, recueilli à Cuba entre 1986 et 2004. Ce graphe a été obtenu notamment par suivi de contacts. Il recense près de 5400 patients et comporte une composante connexe très grande, regroupant presque 2400 patients. La très grande taille de cette composante rend difficile une analyse de sa structure directement au niveau des patients. Nous montrons comment des outils développés pour la visualisation de grands graphes peuvent être appliqués à ce type de réseau d'infection. Ils permettent notamment une exploration hiérarchique de la structure du graphe : un graphe simplifié dont chaque sommet représente un groupe de patients est d'abord proposé. Il résume la connectivité globale du graphe en cachant la structure au sein de chaque groupe. L'analyse peut développer à la demande les groupes pour étudier la structure locale sans se perdre dans le graphe global. Nous montrons comment les visualisations ainsi produites peuvent être utilisées pour étudier divers aspects de l'épidémie (propagation,
effet de l'orientation sexuelle, etc.).
|